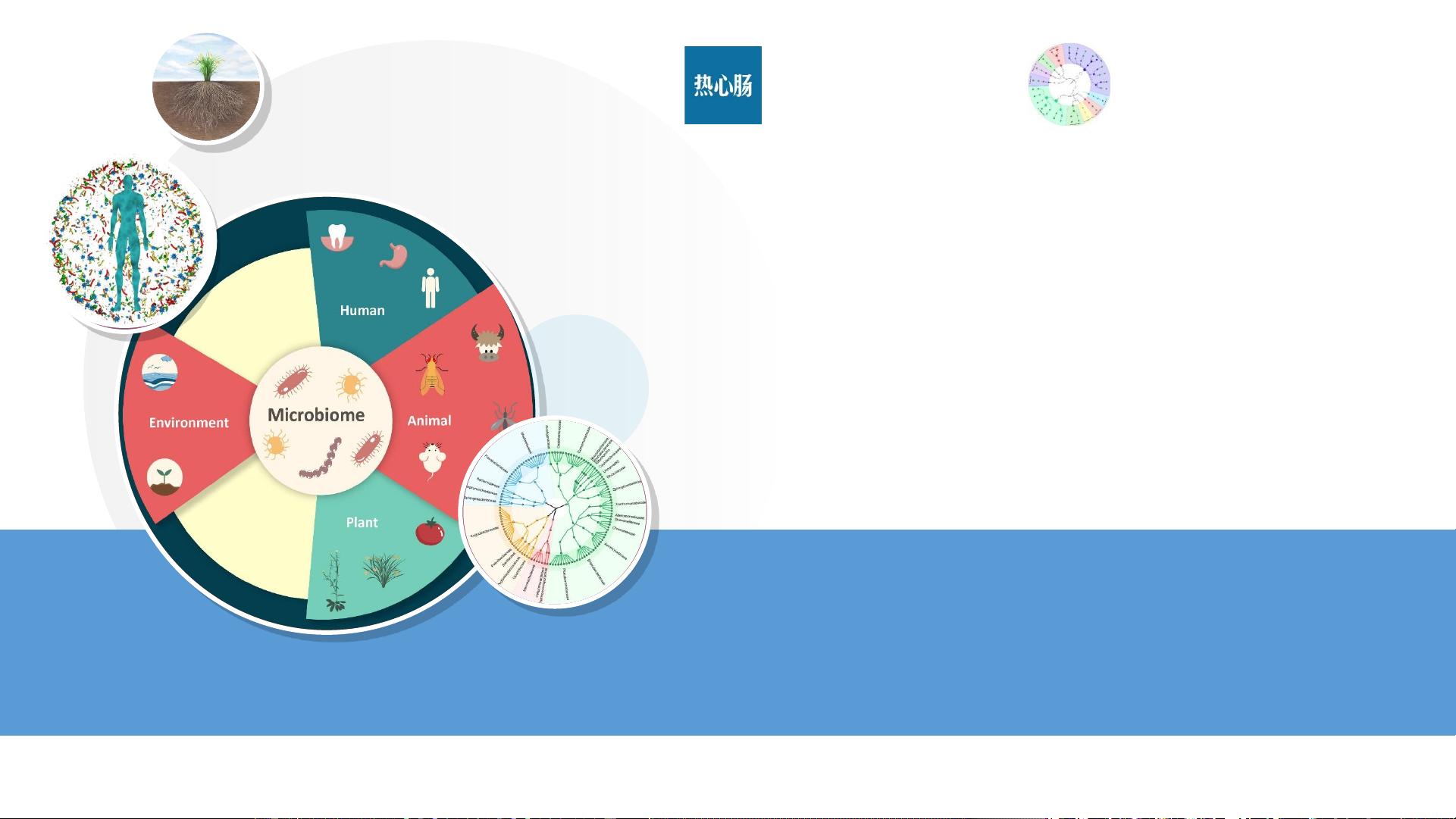
【扩增子分析流程简介】 扩增子分析是宏基因组学研究中一个重要的步骤,主要针对微生物群落的研究,通过测序特定区域(扩增子)的基因,如16S rRNA基因,来揭示样本中的微生物组成和多样性。这种分析流程包括多个阶段,从数据预处理到下游的生物信息学分析。 【硬件需求】 进行扩增子分析通常需要一定的计算资源,包括高性能的个人电脑或者服务器/集群。双显示器可以提高多任务管理效率,而服务器或集群对于大数据量的处理至关重要。操作系统方面,Linux环境和Shell命令行操作是常见的选择,配合R语言和RStudio作为数据分析和可视化的环境。此外,还需要使用像GitForWindows这样的工具来管理和同步代码,以及XShell/PuTTY进行远程服务器访问,Filezilla/WinSCP用于文件传输。 【软件工具】 1. **Shell**: Shell是Linux和Unix系统中的一种命令行接口,用于执行系统命令。 2. **R**: R语言是统计分析和图形绘制的重要工具,RStudio提供了一个集成开发环境。 3. **IDE**: GitForWindows在Windows上提供了Git命令行环境。 4. **扩增子分析软件**: 包括USEARCH和VSEARCH,两者都能处理扩增子数据,USEARCH功能强大但需付费,VSEARCH是开源的替代品。 5. **QIIME 2**: QIIME(Quantitative Insights Into Microbial Ecology)是一个广泛使用的微生物群落分析软件,QIIME 2是其最新版本,提供了一整套分析流程。 6. **辅助工具**: seqkit用于序列操作,csvtk处理表格数据,rush管理并行任务。 7. **差异分析与可视化**: STAMP用于统计检验和可视化差异表达。 8. **网络分析与可视化**: Cytoscape和Gephi用于复杂网络的构建和展示。 9. **图片排版**: Adobe Illustrator用于高质量的图像编辑和排版。 10. **登录服务器工具**: XShell和PuTTY提供SSH连接到远程服务器。 11. **文件传输**: Filezilla和WinSCP用于FTP/SFTP文件传输。 【扩增子分析基本流程】 1. **数据质量控制**: 包括去除接头序列、低质量读段、去除质控失败的样本等。 2. **OTU聚类**: 对比序列并将其聚类为Operational Taxonomic Units (OTUs)。 3. **OTU注释**: 利用数据库将OTUs与已知的微生物分类信息关联起来。 4. **丰度统计**: 计算每个OTU在样本中的相对丰度。 5. **α多样性分析**: 评估单个样本的多样性,包括物种丰富度、均匀度等指标。 6. **β多样性分析**: 比较不同样本间的群落结构差异,例如UniFrac距离和PCoA分析。 7. **差异分析**: 识别不同条件或组间显著差异的OTUs。 8. **网络分析**: 揭示微生物之间的相互作用关系。 9. **结果可视化**: 通过各种图表(如箱线图、PCA图、热图等)呈现分析结果。 【易用的扩增子分析流程】 EasyAmplicon和QIIME 2都是简化扩增子分析的流程。EasyAmplicon提供了一个简单易用的界面,适合初学者。而QIIME 2则是一个全面的分析平台,包含多种预定义的分析工作流,允许用户自定义分析参数,适合更深入的研究。 总结来说,扩增子分析是一个涉及多步骤、多软件工具的过程,需要合适的硬件环境和丰富的生物信息学知识。通过理解并熟练运用这些工具,研究人员可以深入解析微生物群落的结构、功能和动态变化。
剩余58页未读,继续阅读
- 粉丝: 934
- 资源: 316
 我的内容管理
展开
我的内容管理
展开
 我的资源
快来上传第一个资源
我的资源
快来上传第一个资源
 我的收益 登录查看自己的收益
我的收益 登录查看自己的收益 我的积分
登录查看自己的积分
我的积分
登录查看自己的积分
 我的C币
登录后查看C币余额
我的C币
登录后查看C币余额
 我的收藏
我的收藏  我的下载
我的下载  下载帮助
下载帮助

 前往需求广场,查看用户热搜
前往需求广场,查看用户热搜最新资源
- NE555+74LS192+74LS48电子秒表课程设计报告(纯数电实现)
- 基于深度学习的视频描述综述:视觉与语言的桥梁
- 2024年全球干式变压器行业规模及市场占有率分析报告
- 小红书2024新年市集合作方案解析与品牌营销策略
- 基于javaweb的沙发销售管理系统论文.doc
- 毕业设计Jupyter Notebook基于深度网络的垃圾识别与分类算法研究项目源代码,用PyTorch框架中的transforms方法对数据进行预处理操作,后经过多次调参实验,对比不同模型分类效果
- 基于java的扫雷游戏的设计与实现论文.doc
- 基于java的企业员工信息管理系统论文.doc
- 深度视频压缩框架:从预测编码到条件编码的技术革新
- 1221额的2的2的2额


 信息提交成功
信息提交成功





评论0