DNA元基催化与肽计算_第5修订版本1
需积分: 0 154 浏览量
更新于2022-08-08
收藏 8.43MB DOCX 举报
【DNA元基催化与肽计算】这一主题涉及的是生物信息学和计算化学的交叉领域,主要探讨DNA分子在催化反应中的作用以及与蛋白质(肽)计算的关联。在这个第五修订版的提案中,作者着重展示了德塔自然语言图灵系统的性能测试,该系统基于Windows 10操作系统,在联想Y7000笔记本上运行,实现了中文分词的高效处理。
中文分词是自然语言处理中的关键步骤,德塔系统在此方面表现出色,每秒能够处理1630到1650万个中文字符,词库规模达到65000+。其分词算法的准确率高达99.7%以上,语法函数缺失率低于0.3%,并且100%开放源码,用户可以通过API或相关书籍获取和使用。
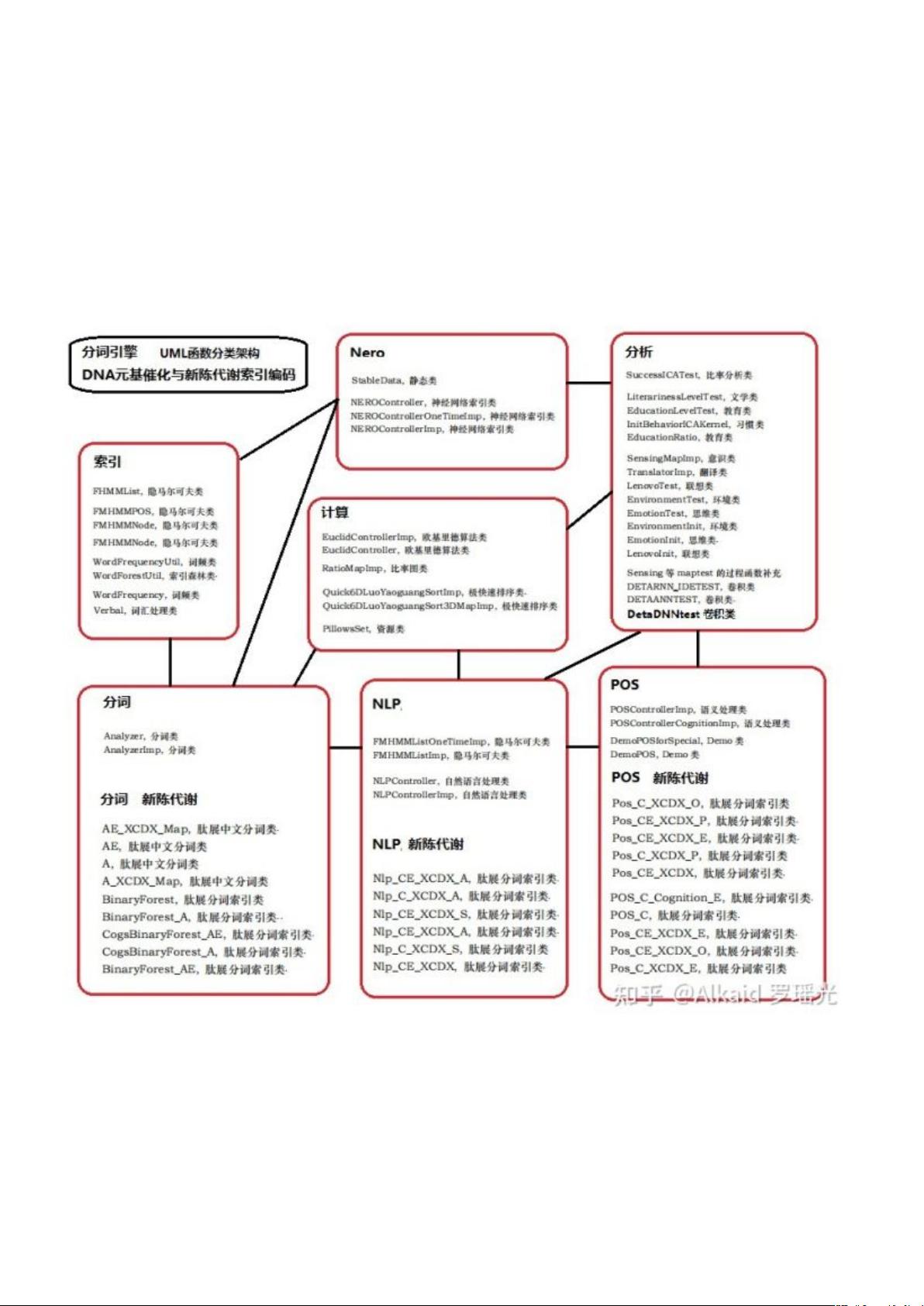
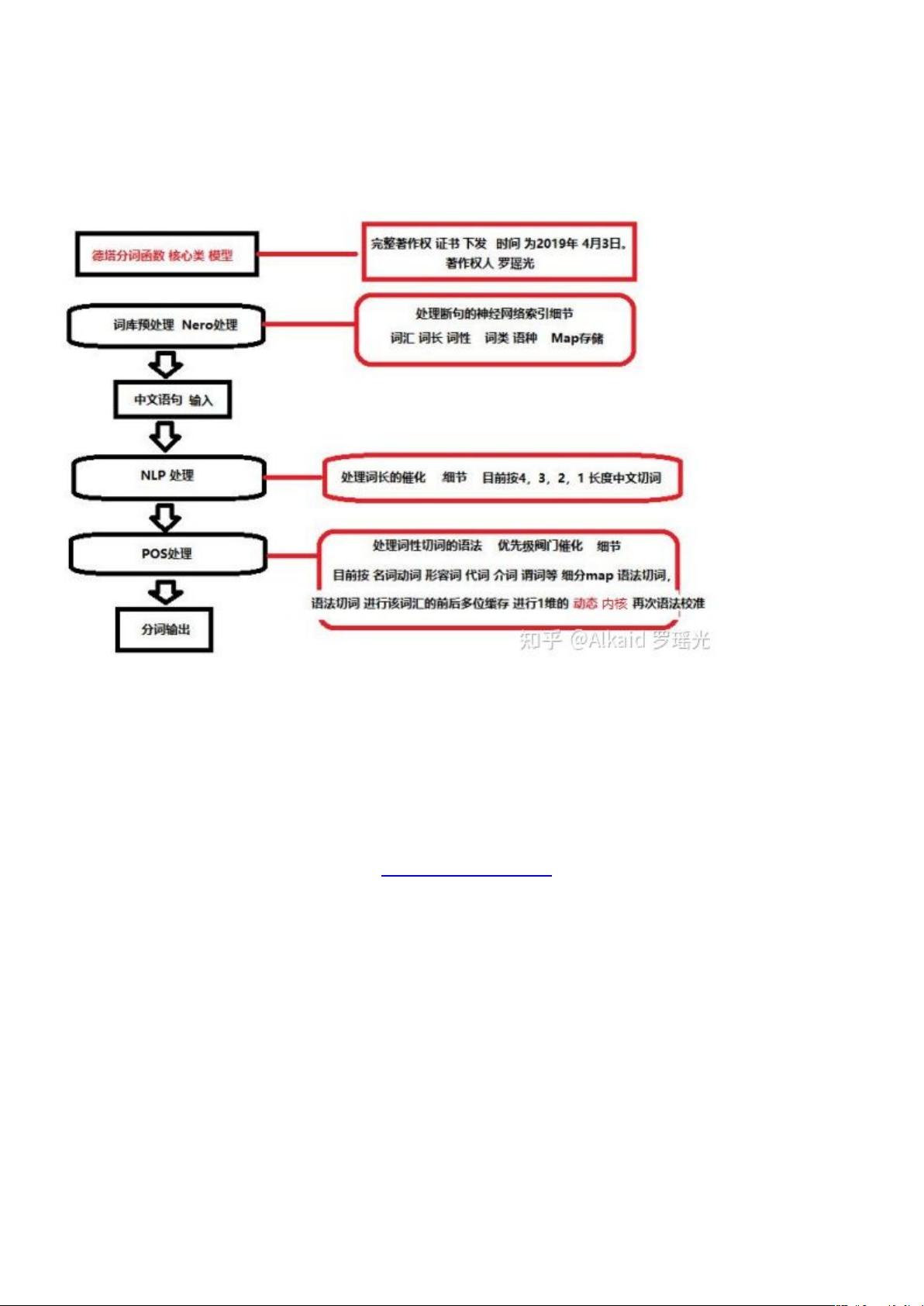
德塔分词引擎采用了基于神经网络的索引字典切割方法,前序遍历词性组合匹配,同时结合文学语法定义进行切词。其优化策略包括细化索引字典、使用频率统计排列优化、动态类卷积遍历关键字优化、文件新陈代谢和词汇匹配细化等。分词过程首先通过对文字逐字遍历索引,然后按长度提取词汇,再进行词性切分。此外,德塔分词还利用关联分类生成的小文件map集进行整体加速,词汇匹配支持多种语言字符集,最大处理长度为4,采用类似CNN卷积的内核计算进行POS识别。
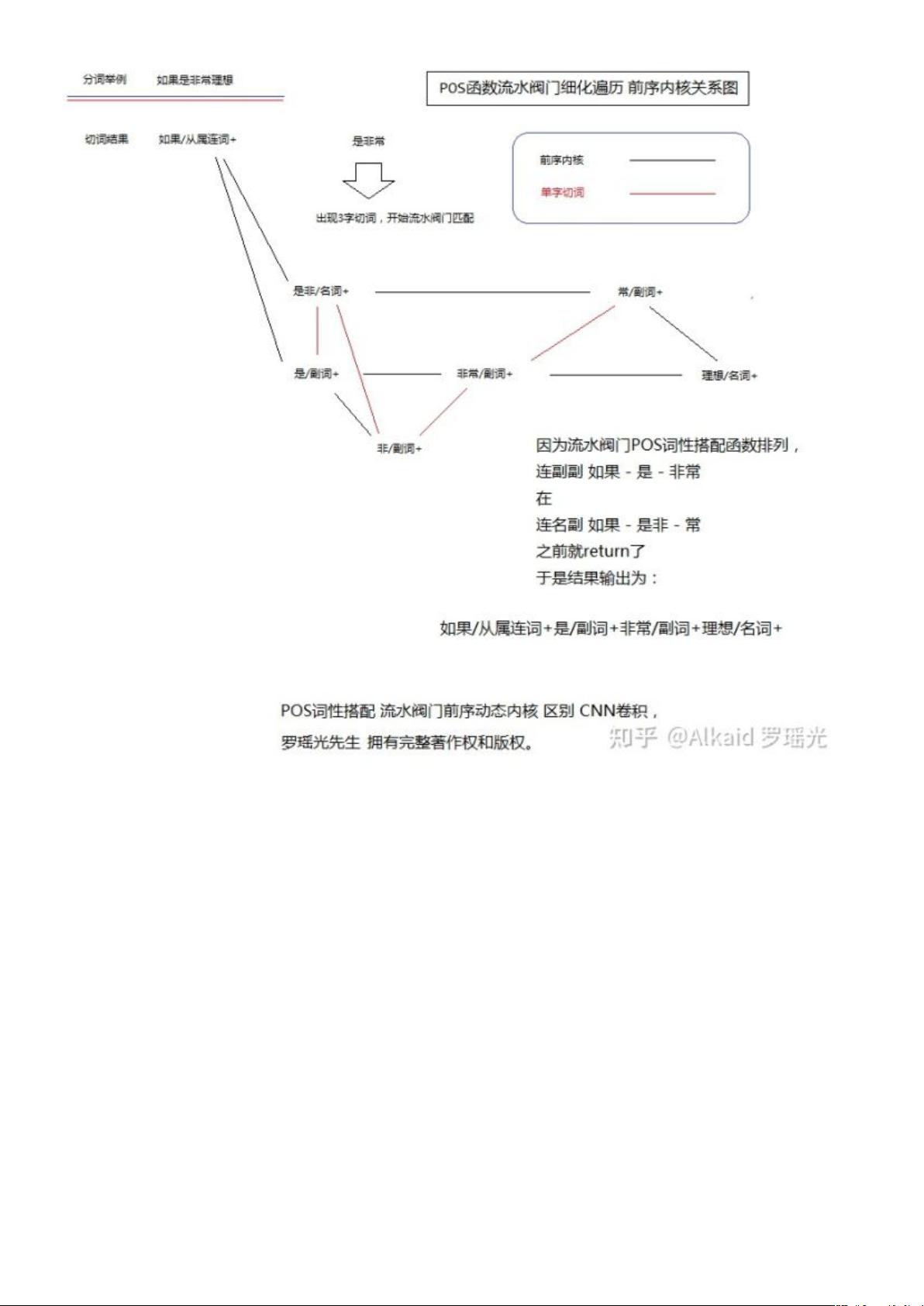
词性切分部分,德塔系统按照4字词、3字词、2字词和单字的顺序,依据词汇的POS搭配语法模式进行归纳,根据文本中POS出现频率进行优化。在排序算法上,德塔分词借鉴了快速排序的思想,并进行了微分催化算子的优化,引入了小高峰左右比对法、波动算子过滤思想和离散条件归纳微分思想。
在分词引擎和相关组件的优化中,德塔系统采用微分催化系统,例如在词汇字典索引上,利用JDK8+的Map对象支持的二分搜索,提高了搜索速度。索引通过细化分类如词长、词类和词性Map来进一步加速搜索。同时,德塔分词的索引Map支持两次组合计算,适应分布式服务器的索引缓存,但作者建议在分布式环境下使用。
【DNA元基催化与肽计算】的研究不仅涵盖了生物学的基础知识,还深入到了计算技术的前沿,特别是在自然语言处理和中文分词算法方面,展现了高效、精准的技术实现。德塔自然语言图灵系统通过一系列优化策略,为中文文本的处理提供了强大工具,具有很高的实用价值和研究意义。
XiZi
- 粉丝: 735
- 资源: 325
最新资源
- python操作arxml.txt
- python操作abaqus.txt
- python操作excel图片.txt
- python操作gitlab.txt
- python操作excel导出图片.txt
- python操作mysql教程pdf.txt
- python操作pdf和ppt.txt
- python操作pdfminer.txt
- python操作pdf文件.txt
- python操作pdf获取文本.txt
- python操作table标签.txt
- python操作ts音频流.txt
- python操作tsc打印机打印.txt
- python操作txt删除行.txt
- python操作word插入图片.txt
- python操作xml导入什么库.txt