cytoscape使用教程
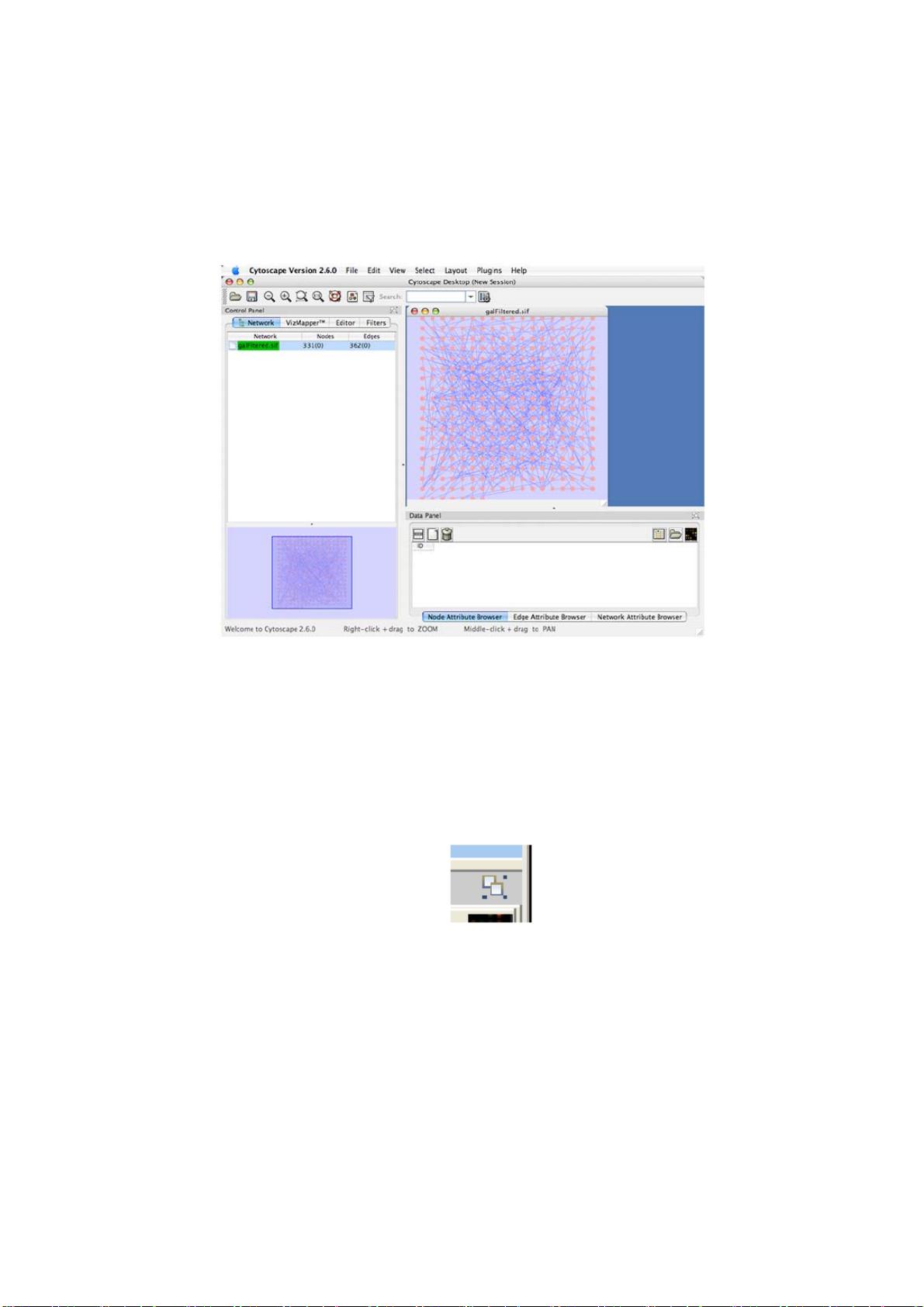
Cytoscape是一款开源的网络可视化和分析软件,最初用于生物学领域,整合分子间相互作用的网络和分子状态信息。Cytoscape支持通过插件扩展,以适应各种附加的计算分析和功能需求。其核心功能包括基础的网络布局、查询以及将数据整合为可视化网络。软件的界面布局清晰,包括菜单栏、编辑、视图、选择、布局、插件和帮助等主要部分。 Cytoscape安装时需要满足系统性能要求,包括足够的内存和栈空间。新版本如2.6包含了多个新特点,例如WebServiceClientManager框架用于整合网络服务客户端,以及动态过滤、标签位置改进和改良的XGMML等功能,以提升软件的执行性和可用性。此外,该版本还修复了许多bug。 Cytoscape的网络是由节点(如分子、因子、蛋白质)和边(代表两点间的交互关系)组成。用户可以通过Cytoscape分析网络数据,并将网络与文件、表型和其他分子状态信息关联。Cytoscape支持与大型数据库的交互,例如蛋白质、DNA以及遗传信息相关的数据库。 Cytoscape的发展受到了多个研究机构和公司的支持,例如Institute for Systems Biology、加利福尼亚大学圣地亚哥分校、纪念斯隆-凯特琳癌症中心、巴斯德研究所、安捷伦科技、加利福尼亚大学旧金山分校等。软件遵循GNU LGPL协议,允许用户免费使用,并且可以在线获取其开源库和相关的详细信息。 用户在使用Cytoscape时,可以使用其File(文件)、Edit(编辑)、View(视图)、Select(选择)、Layout(布局)、Plugins(插件)和Help(帮助)等功能来完成网络的构建、编辑、分析等工作。该软件支持多网络选择和保存,标签位置的优化,以及会话的内存保存等功能。 Cytoscape的主题和样式支持用户的个性化需求,用户可以根据自己的偏好选择或创建不同的视觉样式。同时,Cytoscape可以与外部服务如NCBI Entrez、Gene BioMart进行集成,以支持网络数据的下载和ID转换等功能。 Cytoscape为研究人员提供了一个强大的平台,以便他们能够更好地可视化和分析复杂的网络数据。由于其开源的性质,用户可以免费使用和修改软件,同时也可以参与到软件的开发和改进中,通过社区贡献新的插件或反馈错误和改进建议。




剩余27页未读,继续阅读

 yiyang20110130a2017-01-13很详细的介绍,可以参考。
yiyang20110130a2017-01-13很详细的介绍,可以参考。
- 粉丝: 1
- 资源: 2
 我的内容管理
展开
我的内容管理
展开
 我的资源
快来上传第一个资源
我的资源
快来上传第一个资源
 我的收益 登录查看自己的收益
我的收益 登录查看自己的收益 我的积分
登录查看自己的积分
我的积分
登录查看自己的积分
 我的C币
登录后查看C币余额
我的C币
登录后查看C币余额
 我的收藏
我的收藏  我的下载
我的下载  下载帮助
下载帮助

 前往需求广场,查看用户热搜
前往需求广场,查看用户热搜最新资源
- 基于java的校园招聘系统的设计与实现.docx
- 基于java的协同过滤算法商品推荐系统的设计与实现.docx
- 基于java的新冠物资管理系统的设计与实现.docx
- 基于小程序的高校毕业管理系统小程序源码(小程序毕业设计完整源码+LW).zip
- 基于java的新冠物资管理的设计与实现.docx
- 基于java的学生心理压力咨询评判系统的设计与实现.docx
- 基于java的学生信息管理系统的设计与实现.docx
- 基于java的学生成绩分析和弱项辅助系统的设计与实现.docx
- 基于java的综合小区管理系统的设计与实现.docx
- 基于群智能体优化算法的无人机集群路径规划算法,MATLAB仿真,可生成包括每架无人机路径、高度、威胁、转角、迭代次数等,有代码讲解,运用蜣螂算法、豪猪算法、蚁群算法来对无人机集群路径规划,分布式规划
- 基于小程序的鲜花销售源码(小程序毕业设计完整源码+LW).zip
- Drivedroid(重装系统)安全下载.apk
- 使用 Docker 和 Flask 集成 PostgreSQL 数据库并部署应用程序
- MATLAB基于粒子滤波的锂离子电池寿命(SOH)预测 附详细资料 使用美国宇航局 NASA 埃姆斯研究中心的 4 组锂离子电池老化试验数据进行 matlab 仿真进行寿命预测,实验证 明粒子滤波能够
- 计算机专业100套毕业设计(内含JAVA源码)
- 基于小程序的语言课学习系统的设计与实现源码(小程序毕业设计完整源码).zip


 信息提交成功
信息提交成功