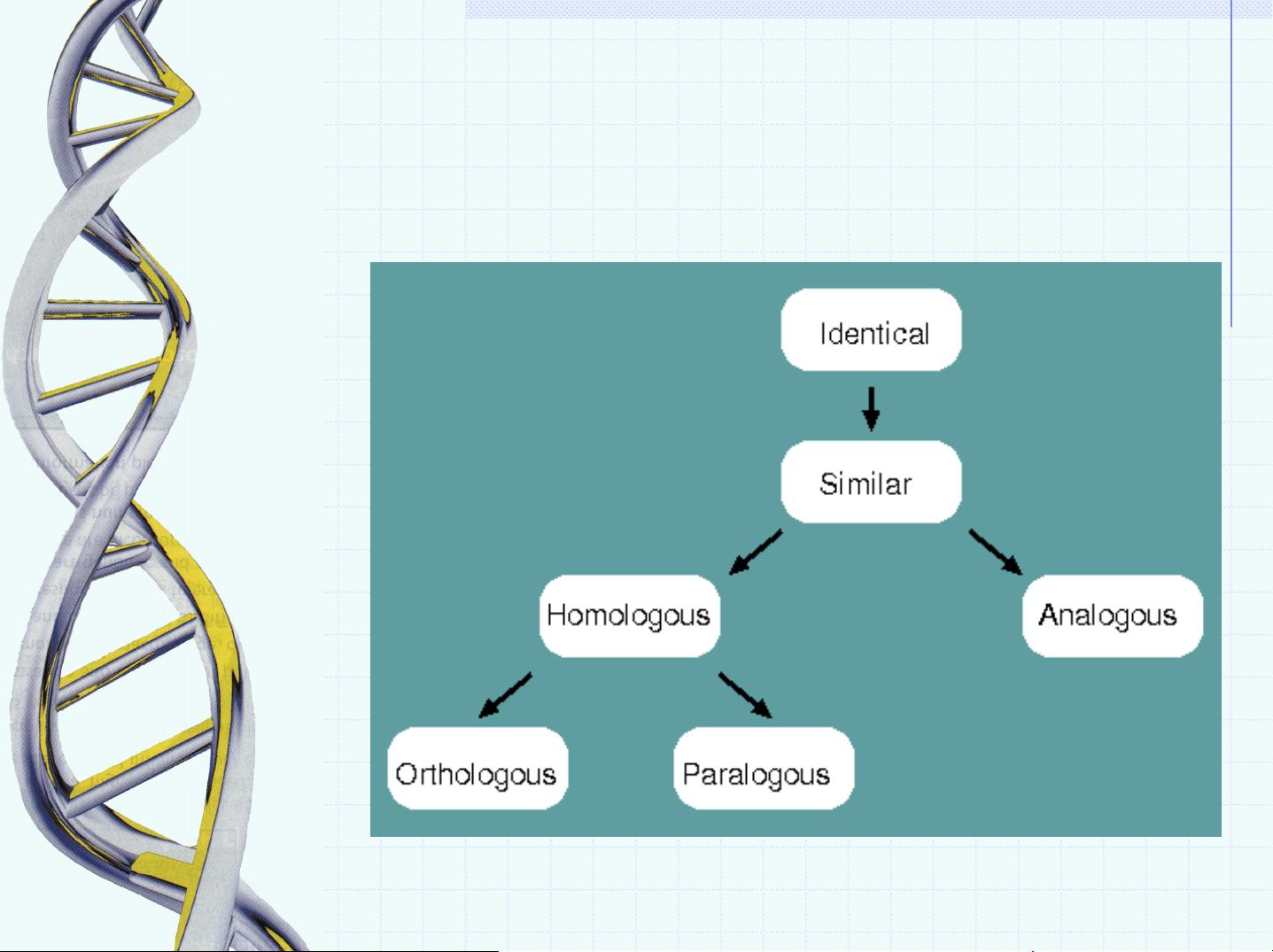
《BLAST:强大的序列比对工具与生物信息学分析》 BLAST,全称为Basic Local Alignment Search Tool(基本局部比对搜索工具),是由Altschul SF等人在1990年提出的,作为生物信息学领域中最广泛使用的序列比对工具之一,其设计初衷旨在解决生物学研究中日益增长的序列数据比对需求。BLAST不仅融合了动态规划算法的精确性和间接启发式算法的高效性,还在统计学基础上实现了快速准确的数据库搜索,极大地推动了分子生物学、遗传学和蛋白质组学等领域的研究进展。 ### 同源与相似性的概念区分 在生物信息学中,同源性和相似性是两个经常被提及但含义截然不同的概念。相似性指的是序列之间在特定区域的相似程度,通常通过序列匹配率或特定度量标准来量化;而同源性则是指两个序列在进化历史上拥有共同祖先的关系,是一种基于历史演化的判断。例如,两个蛋白质序列如果在多个位点上具有相同的氨基酸,它们就表现出较高的相似性;但如果这两个序列在进化过程中源自同一祖先,那么它们就是同源的。 ### 序列比对类型及算法 序列比对分为全局比对和局部比对外,前者关注整个序列之间的最佳匹配,适用于分析长度相近、整体结构相似的序列;后者则更侧重于寻找序列中局部区域的相似性,尤其适用于那些仅在某些关键片段有相似性的序列比对。在算法层面,动态规划算法虽然能提供最精确的比对结果,但由于计算复杂度高,难以应对大规模数据处理。相比之下,间接启发式算法如BLAST,通过索引字串等技术显著提升了搜索效率,成为生物信息学分析的首选工具。 ### BLAST的应用场景与优势 BLAST的主要目标是帮助科学家识别未知序列的功能,寻找多基因家族成员,确定蛋白质或核酸中保守区域,以及在组装测序反应时定位重叠区域。具体到操作层面,BLAST能够: - **识别未知序列**:当面对未知序列时,BLAST能够快速地在已知数据库中找到相似序列,从而推测其可能的功能和来源。 - **多基因家族分析**:通过比对序列,BLAST有助于发现和研究基因家族中不同成员之间的关系,这对于理解基因组结构和进化至关重要。 - **蛋白质功能预测**:BLAST能够找出与查询序列相关的蛋白质,通过对这些已知蛋白质的研究,推测查询序列可能的生物学功能。 - **保守区域鉴定**:通过比对,BLAST可以帮助识别蛋白质或核酸序列中高度保守的区域,这些区域往往与特定的生物学功能密切相关。 - **序列拼接**:在DNA测序项目中,BLAST能够辅助识别并合并来自不同测序反应的重叠片段,最终构建完整的基因序列。 ### BLAST的不同版本 - **Gapped BLAST**:这是BLAST的一个改进版本,允许在比对中存在缺口,这意味着序列中的缺失或插入也可以被正确处理,提高了比对的灵活性和准确性。 - **PSI-BLAST (Position-Specific Iterated BLAST)**:这是一种迭代式的比对方法,通过多次迭代逐步构建位置特异的评分矩阵,用于后续的数据库搜索,从而提高特定位置匹配的精确度。这种方法特别适合于检测远亲关系的序列。 BLAST作为生物信息学领域内不可或缺的工具,其在序列比对、同源搜索等方面展现出的强大功能,不仅极大地方便了科研人员的数据分析工作,也促进了生命科学领域的深入探索。随着生物数据的爆炸式增长,BLAST及其后续的优化版本将持续发挥着核心作用,助力生物学家们解开生命奥秘的关键线索。

剩余61页未读,继续阅读

- 粉丝: 0
- 资源: 2
 我的内容管理
展开
我的内容管理
展开
 我的资源
快来上传第一个资源
我的资源
快来上传第一个资源
 我的收益 登录查看自己的收益
我的收益 登录查看自己的收益 我的积分
登录查看自己的积分
我的积分
登录查看自己的积分
 我的C币
登录后查看C币余额
我的C币
登录后查看C币余额
 我的收藏
我的收藏  我的下载
我的下载  下载帮助
下载帮助

 前往需求广场,查看用户热搜
前往需求广场,查看用户热搜最新资源


 信息提交成功
信息提交成功